第Ⅰ部 生命科学データ別にファーストアクションを学ぶ
第1章 ゲノム
河野暢明
de novo ゲノムシークエンスで新たにゲノム配列を決定した際,まず行われるファーストアクションは決定されたゲノム配列の質評価である.これはそのゲノムをどのような研究目的に基づいて決定されたかに依らず,ほとんどのケースで必要最低限求められる初期解析となる.本章ではゲノムアセンブリと遺伝子アノテーションまでが完了したドラフトゲノムを手に入れた地点をスタートとし,ゲノム配列の質を評価するための具体的な定量指標について説明するとともに,得られたゲノム配列を基に初期段階で実施できる解析の例をいくつか紹介する.これによりwet/dry研究者関係なく,誰でもゲノムプロジェクトのファーストアクションが可能になる世界をめざす.
<本章で目標とする図・アクション>① ゲノム統計値(assembly-statsによる描画)
② コンタミネーション評価(blobplot)
機器 : iMac(24インチ2023,チップApple M3,メモリ16 GB,macOS Sonoma 14.5)
データ :
アセンブリファイル(アクセッション番号:GCA_021605075.1,assembly.fasta),シークエンスリード(アクセッション番号:DRR287755,read.fastq.gz),taxonomyデータ(taxdump.tar.gz),タンパク質データ(Reference_Proteomes_2024_05.tar.gz)
言語 : Python,Perl,シェルスクリプト
Pythonパッケージ : sys,subprocess
コマンドラインツール : assembly-stats,SeqKit v2.9.0,gVolante,bwa,samtolls,diamond
はじめに
新規にゲノムを決定する際,実験および解析の手順は実験,バイオインフォマティクス解析,ゲノム評価の大きく3つの段階に分けられる.実験段階では,サンプルからのゲノム抽出,ライブラリの調製,そしてゲノムシークエンスが行われる.次に,バイオインフォマティクス解析として,得られたシークエンスリードを基にしたゲノムアセンブリ,さらに遺伝子予測およびアノテーションが実施される.そして最終的に,ゲノム評価でゲノムの質評価および初期解析が行われる.この一連のプロセスにおいて,特に実験およびバイオインフォマティクス解析は対象とする生物や用いるシークエンス法によってプロトコールや解析手順が大きく異なるため,本章では詳細な解説を控える.これらに関しては「誰でも再現できるNGS『前』サンプル調製プロトコール」1)が参考になる.ここでは,ゲノムアセンブリと遺伝子予測・アノテーションが完了し,ドラフトゲノムが得られた段階を起点とし,ほぼすべてのゲノムプロジェクトで共通して行われるゲノム評価を初期解析(ファーストアクション)として紹介する.ゲノム評価で実施される主な解析は,シークエンスリードのアセンブリ評価である.ゲノムアセンブリが完了した後,その結果がどの程度優れているかを評価することは重要である.その指標の1つとして用いられるのがN50である.N50とは,ゲノムアセンブリ結果におけるコンティグ(連結配列)の結合度合いを示すものである.この値が高ければ高いほど,アセンブリされたゲノムがより完全に繋がっていることを意味する.ただし,N50が高くとも,ゲノム内に含まれる情報に偏りがある場合,そのゲノムの利用価値は大きく損なわれる.これを補完する指標として,BUSCO(Benchmarking Universal Single-Copy Orthologs)2)がある.BUSCOは,ある分類群に共通して保存されているシングルコピーのオルソログ遺伝子セットを用い,アセンブリされたゲノムにそのオルソログ遺伝子セットがどれだけの割合で含まれているかを測定するものである.この指標は100%を最高値とし,高い値ほどアセンブリの網羅性が高いことを示す.こうして算出されたゲノム統計値は,論文中でテーブル形式として数値のみが示されることもある(目標図1.1A).しかし,目標図1.1Bのように可視化することで,ゲノムプロジェクト全体を視覚的に把握しやすくなり,評価や比較が容易になる.
もう1つ重要な解析として,ゲノムアセンブリ結果における純度評価がある.単一サンプルから抽出されたゲノムDNAであっても,微小生物由来のDNAや実験操作中のコンタミネーション(汚染)が混入する可能性がある.これを検出するには,各コンティグがどういった分類群由来なのか,GC含量やカバレッジ(リードの被覆率)は全体と同等かなどを計算することで,異質なコンティグを特定することが求められる.さらに,これらのパラメータを可視化することによって,当該ゲノムアセンブリにおけるコンタミネーションが少ないことを証明する図として活用することも可能である(目標図1.2).
そこで本章ではまずゲノム統計値の算出方法および,ゲノム統計値を可視化するためのツールであるassembly-statsの使用方法について①で説明する.次にコンタミネーションの可能性があるコンティグを特定するための可視化手法として,blobplotの作成と解析手順についても②で紹介する.なお本章で例示している図表データは我々の論文3)で発表されたクモ(Caerostris dawrini)のアセンブリデータを基に作られており,アセンブリファイルはassembly.fasta,アセンブリに用いたシークエンスリードはread.fastq.gzとした.これらのデータは,NCBIデータベースでアクセッション番号を検索することで入手できる.
①ゲノム統計値
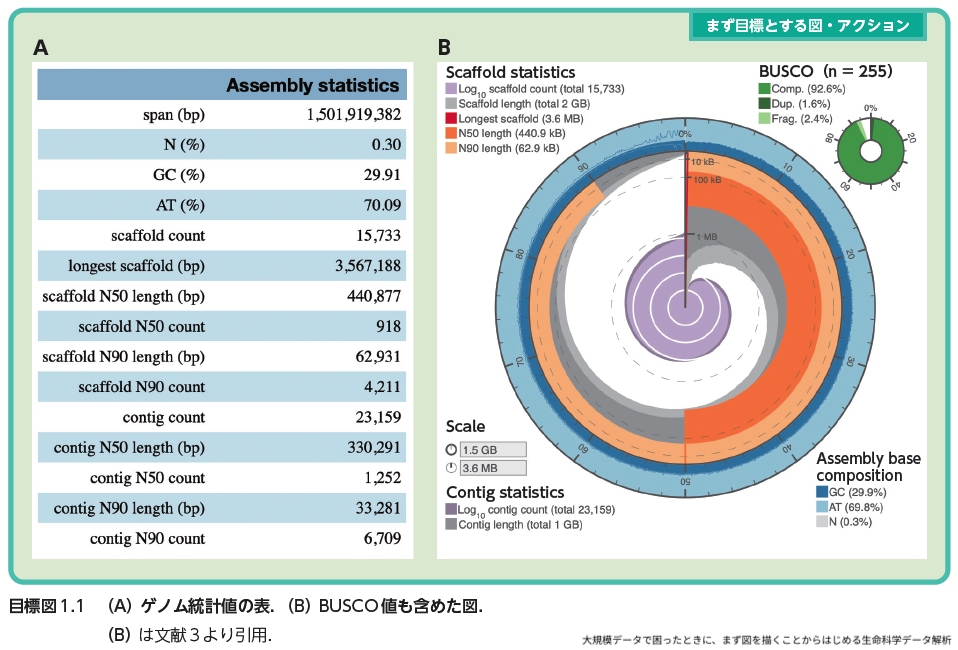
ゲノムの質を評価する際に広く用いられているゲノム統計値の表(目標図1.1A)とBUSCO値も含めた図(目標図1.1B)を例示する.どちらも最近のゲノム論文でよく見かけるassembly-stats4)による描画結果である.
1.1 ゲノム統計値の計算
ゲノム品質を定量評価するために,ゲノム統計値とBUSCOの計算を行う.ゲノム統計値の計算はSeqKitを用いるのが最も簡便に行える.SeqKit5)は配列操作するためのツールで,fastaやfastqフォーマットのファイルを高速に低メモリで扱うことができる.SeqKitはウェブサイト 6)へアクセスし,最新版のバイナリファイルをダウンロード,解凍して実行権を与えるだけで使えるようになる.2024年11月現在ではv2.9.0が最新であったため,そのファイルをダウンロードして計算を進めた.



SeqKitを実行すると,バージョンと使えるコマンド一覧が表示される.紙面の関係で今回関連するコマンド付近のみを掲載しているが,seqkitは基本的にCommands for Basic Operationとして列挙されているコマンドと一緒に使用する.ゲノム統計値はstatsというコマンドで計算することができる.使用方法は以下の通りである.

seqkit statsで使えるさまざまなオプション(flags)と出力されるデータを見てみる.
ご覧ください
文献
- 「誰でも再現できるNGS「前」サンプル調製プロトコール」(鹿島 誠,他/編),羊土社(2024)
- Manni M, et al:Mol Biol Evol, 38:4647-4654, doi:10.1093/molbev/msab199(2021)
- Kono N, et al:Open Biol, 11:210242, doi:10.1098/rsob.210242(2021)
- sanger-pathogens:assembly-stats https://github.com/sanger-pathogens/assembly-stats(2025年1月閲覧)
- Shen W, et al:PLoS One, 11:e0163962, doi:10.1371/journal.pone.0163962(2016)
- SeqKit - Ultrafast FASTA/Q kit:Download https://bioinf.shenwei.me/seqkit/download/(2025年2月閲覧)

