- [SHARE]
- ツイート


はじめに
近年,ゲノム編集ツールを用いた,標的遺伝子の精密な改変が可能となってきた.遺伝子ノックアウトや遺伝子ノックインに加えて,一塩基レベルでの改変も効率的になりつつある.加えて,DNA切断ドメインの替わりに,さまざまなエフェクターを連結した新しいツールが開発され,その応用技術の開発は留まるところを知らない.ここでは,ゲノム編集の原理と応用技術について解説し,さまざまな分野での可能性について紹介する.
DNA二本鎖切断の誘導と修復を介した遺伝子改変
ゲノム編集は,ZFN(zinc-finger nuclease)やTALEN(transcription activator-like effector nuclease)などの人工制限酵素あるいはCRISPR-Cas9(clustered regularly interspaced short palindromic repeats-CRISPR associated protein9)のようなRNA誘導型DNA切断酵素を利用して,標的DNAの塩基配列を改変する技術である1).人工制限酵素は,タンパク質のDNA結合ドメインを標的配列に応じて作製するのが煩雑であったが,2012年にCRISPR-Cas9が開発され2),短いRNA(ガイドRNA)とそれに結合するCas9ヌクレアーゼによって,研究者であれば簡便にゲノム編集を行うことが現在可能となっている.最近これらの標的DNAを切断する人工酵素やRNA-タンパク質複合体はゲノム編集ツールとよばれている.
ゲノム編集では,前述のゲノム編集ツールを細胞や受精卵・胚や個体へ導入し,標的DNAへ特異的なDNAの二本鎖切断(double-strand break:DSB)を誘導する.DSBは細胞にとって有害であるため,細胞に備わるDSB修復機構によって修復されるが,この過程で遺伝子の改変が行われる.DSBの修復には複数の修復経路が関与し,それぞれの修復経路の特性を利用してさまざまなタイプの遺伝子改変を実行することが,現在可能となっている(図1).DSBの多くは,末端をつなぎ合わせる非相同末端結合(non-homologous end-joining:NHEJ)修復によって修復される.NHEJ修復ではDSB末端が削られないように保護され,元通りの塩基配列につなぎ合わされる.しかし,正確に修復が行われた場合は標的配列が現れ,残存するゲノム編集ツールによって再びDSBを誘導される.一般に,NHEJはエラーの起こりやすい修復経路であるため,くり返しの修復過程においては短い欠失や挿入などの変異(インデル変異)が導入される.インデル変異が遺伝子のコード領域に導入されると,アミノ酸の情報に変化が生じ,多くの場合,遺伝子の機能が破壊される(遺伝子ノックアウト).またNHEJ修復の際に,ドナーDNAを共導入しておくことによって,DSBの起こった箇所に外来DNAが挿入される(遺伝子ノックイン).NHEJを利用した遺伝子ノックインは,さまざまな細胞種や生物種でその効率は高いものの,正確性に欠ける・挿入の方向性が選べないという問題があったが,CRISPRを利用したHITI法によって正確性かつ効率的なノックインが可能となってきた3).
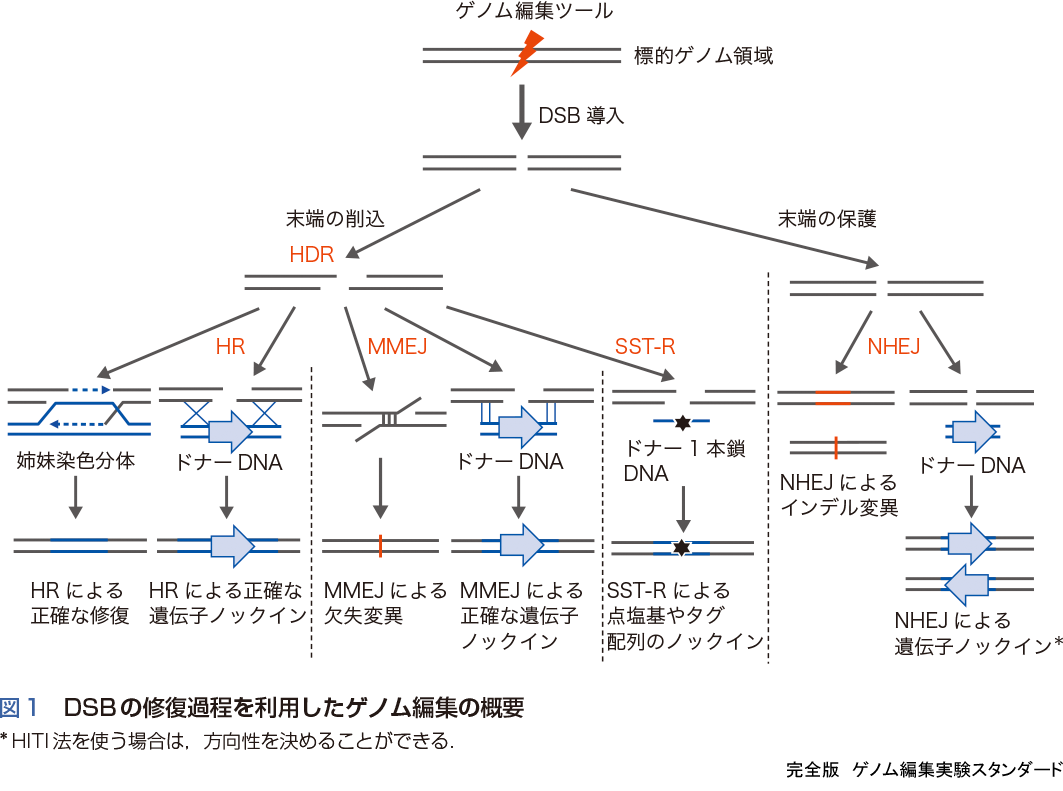
DSB末端の保護によるNHEJ修復に対して,DSB末端の削り込みを利用した複数の修復経路〔相同組換え(HR)やマイクロホモロジー媒介末端結合(mocrohomology-mediated end-joining:MMEJ)修復,一本鎖DNA標的修復(single-strand template-repair:SST-R〕が知られている.これらの修復経路は,相同配列を利用する修復であることから相同配列依存的修復(homology-directed repair:HDR)とよばれ,片方の鎖が削り込まれて一本鎖として凸出した配列を利用して,DSBを修復する.HR修復は,姉妹染色分体の修復に利用される正確性の高い修復経路であるが,細胞周期の中期S期からG2期において活性があるため,増殖する細胞において利用される.この経路を利用すると,切断箇所の両側の比較的長い配列を有するドナーベクターを調製し,共導入することによって,DSB箇所へシームレスに外来DNAを挿入できる.一方,分化した神経細胞のような非増殖細胞ではHR活性が低く,HR修復を利用した遺伝子ノックインは困難である.MMEJは,比較的短い相同配列(数十塩基から数百塩基)を利用した修復経路であり,細胞内のDSBがこの経路で修復されると多くの場合欠失変異が導入される.正常な細胞において,MMEJ活性は細胞周期のG1期からS期はじめの長い期間に活性がみられるため,この経路によって修復される頻度も高い.このようにMMEJによる修復は欠失変異を導入するので,正確な修復とは言えないが,この経路をうまく利用すると効率的な遺伝子ノックアウトが可能である.例えば,ゲノム編集ツールによる切断後の欠失によって,確実にフレームシフトやストップコドンが出現する標的配列を選ぶことができる.また,MMEJを介した遺伝子ノックインでは,ドナーDNAに用いる相同配列は数十塩基程度で十分であり,ドナーDNAの構築が簡便である(Ⅰ-5).さらに,SST-Rは詳細な修復メカニズムは不明であるが,この方法を利用することで一本鎖オリゴDNA(ssODN)や長鎖一本鎖DNA(long ssDNA)を用いて,一塩基改変(SNP導入),タグ配列の挿入などを細胞や個体レベルで実現できる.
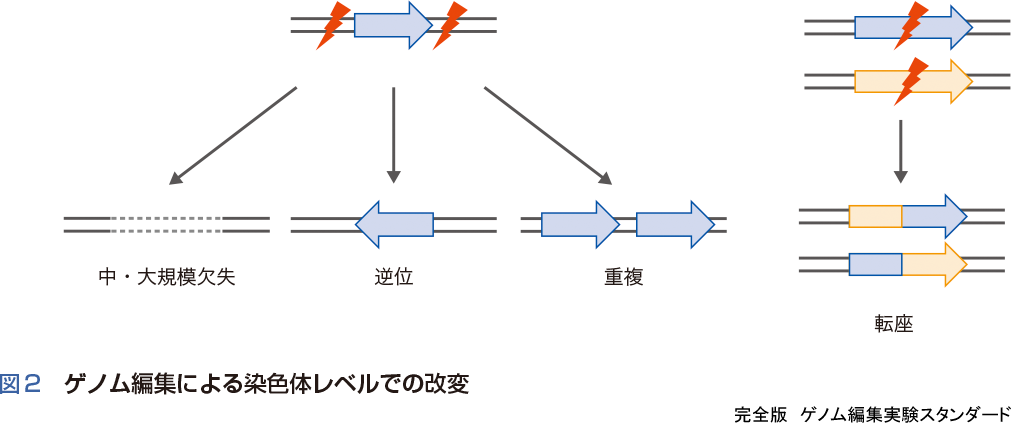
前述の一カ所を切断する方法に加えて,複数箇所を同時に切断することによって中規模から大規模な欠失や染色体再編などを行うことも可能である(図2).複数箇所の改変で利用する修復経路はケースバイケースであるが,同一染色体上の2カ所の標的配列をゲノム編集ツールで切断することによって,大きな欠失や逆位,重複なども可能である.2つの異なる染色体をそれぞれ同時に切断することによって,がん細胞でみられる転座の誘導が細胞や個体のレベルで報告されている.
DNA認識・結合ドメインを利用した新しいゲノム編集技術の体系
ZFNやTALENなどのゲノム編集ツールは,人工制限酵素として高い特異性を有する酵素を開発するために進められてきた.これに対してCRISPR-Cas9は,細菌の有する獲得免疫システムを利用した方法であり,標的とするDNAに特異的に結合するガイドRNAを利用している.両者とも細菌が進化させてきた防御システムを利用している点で非常に興味深い.ゲノム編集ツールは,標的DNAの塩基配列を認識するシステム(DNA認識・結合ドメイン)にDNA切断ドメインを融合した技術として発展してきた.このことは,標的DNA認識・結合ドメインにさまざまな機能ドメインを融合することで新しい技術へ発展可能であることを意味している.すでに図3に示すようにゲノム編集ツールで使われているzinc-finger,TALE,CasヌクレアーゼやdCas(ヌクレアーゼ不活性型Cas)にさまざまな機能ドメインを連結した人工のDNA制御システムが開発されている.特にdCas9に転写因子の活性化あるいは抑制ドメインを連結した人工の転写調節因子を利用した転写調節システム(佐久間らのコラム),DNAやヒストンの化学修飾酵素を連結した人工のエピゲノム調節因子(Ⅱ-6),脱アミノ化酵素を利用した人工デアミナーゼ(Ⅱ-5)などが,人工酵素として開発が進行している.さらに最近,トランスポゼースと複合体を形成する新しいタイプのCRISPRが発見され,遺伝子ノックインへの適用性が示された4).加えて,蛍光遺伝子を連結させたゲノム編集ツールを利用したイメージング技術も開発が競って進められている.PPR(pentatricopeptide repeat)モチーフはRNAの塩基配列特異的に結合するドメインをもつタンパク質として植物から発見され,国産ツールとして期待されている.また,CRISPRの中にはガイドRNA依存的に標的RNAに特異的に結合するタイプ(Cas13aなど)が見つかっている.これらRNAの認識・結合ドメインは,細胞内外の標的RNAの標識や検出技術に利用されている.このような状況から,ゲノム編集は,単に核酸を切断するための技術ではなく,DNAやRNAの認識・結合ドメインを利用した広範な技術を意味する技術へ展開している.組合わせのアイディア次第で,さらに新しい技術が生み出されることが期待される.
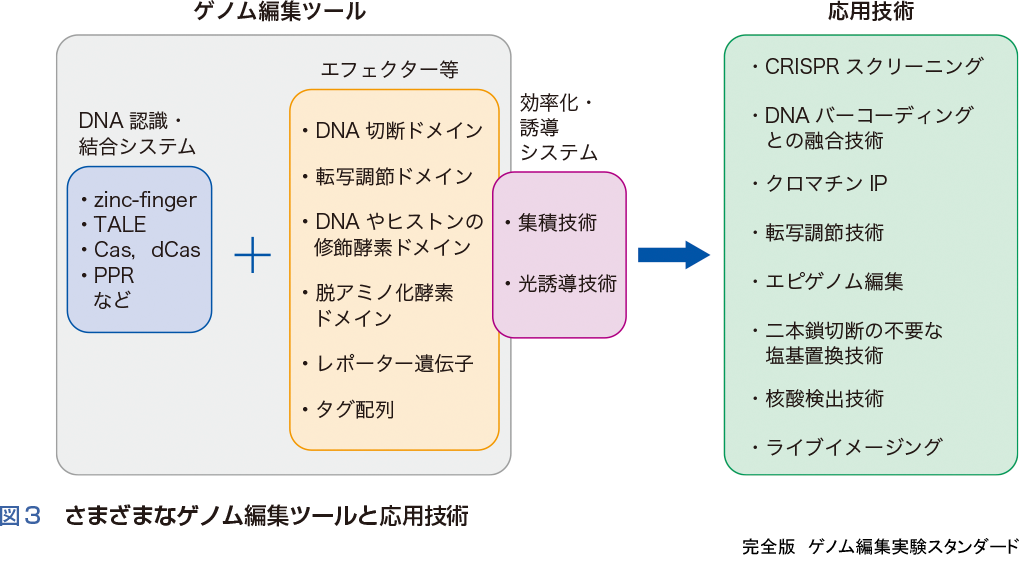
DNAやヒストンの修飾状態(エピゲノム)は遺伝子の転写調節に重要であるが,前述の人工のエピゲノム調節因子を利用してエピゲノム状態を改変することが近年可能になってきた.この技術はエピゲノム編集とよばれ,標的遺伝子のDNAのメチル化やヒストンのアセチル化のレベルを調節することが可能である(Ⅱ-6).さらに,エピゲノム修飾因子を含むさまざまなエフェクターを標的に集積する技術(Sun tagシステム5)やSAMシステム6))が開発され,高効率的な制御を実現するシステムとして進んでいる.
ゲノム編集ツールを利用した発展技術として注目されるのがCRISPRスクリーニングである(Ⅱ-4)7).この方法はこれまで,ゲノム編集が単一あるいは複数でも限られた数の標的遺伝子を対象として解析するという戦略から,機能的な遺伝子や領域を全体から探索するという逆の戦略がCRISPRライブラリーによって可能となってきた.この技術は,すべての遺伝子に対するガイドRNAを容易に作製することができるCRISPR-Casではじめて可能になったものであり,今後この技術を利用した機能遺伝子の探索が加速するであろう.特に,創薬ターゲットの遺伝子探索には必須の技術となる可能性がある.
DNAバーコーディングは,短いタグ配列を指標として生物の進化系統を探る方法として利用されてきた.最近この方法とゲノム編集を融合した技術によって細胞系譜を追跡する方法が開発されている.GESTALT(genome editing of synthetic target arrays for lineage tracing)とよばれる代表的な方法8)は,CRISPRの人工の標的配列をタンデムに連結したターゲットアレイを細胞や個体に導入し,そこから生み出されるさまざまな変異型アレルを系統的に追跡することによって,発生での細胞系譜や複雑な組織・器官での細胞の祖先を追跡する方法である.
この他にも,血液や唾液などの生体試料から,短時間かつ簡便にウイルスや細菌,がんの変異などを検出する技術(SHERLOCK法9)やDETECTR法10).西増のコラム参照),CRISPRを利用したChIP法(Ⅲ-2)など,続々と新しい技術が生み出されている.
ゲノム編集の基礎から応用での可能性
ゲノム編集の魅力は,これまで改変が困難であった生物種において狙って遺伝子改変が可能な点にある.すでにゲノム情報が解読され,遺伝子導入法が確立されている生物種であれば,原理的に標的の遺伝子を選んで改変することが可能である.特にCRISPR-Cas9は,簡便,高効率かつ安価(基礎研究では)に利用できることから,基礎研究においてはすでに基本ツールとなっている.以前は,変異体を扱うことが困難な生物種はモデル生物と言い難かったが,ゲノム編集の開発によって,モデル動物という用語は意味をなさなくなってきた.本書では,さまざまな生物種におけるゲノム編集の詳細が,各章において専門の研究者が原理と実験手法を紹介されている.本書で扱っていない生物種においても,今後続々と基礎研究のために遺伝子ノックアウトがさまざまな生物種を対象とした基礎研究で報告されるであろう.CRISPR-Cas9を利用できれば遺伝子ノックアウトが実現する一方,遺伝子ノックインや塩基レベルでの改変をすべての生物種で遺伝子座に関係なく効率的に行うことは現時点では困難である(さまざまな生物種において改良され高効率にはなってきているが).遺伝子ノックインの効率化においては,標的遺伝子座のエピゲノム状態も重要であり,利用する細胞株や細胞種,個体であれば発生段階によっても大きく異なる場合もある.今後は,塩基配列の情報,DNAやヒストンの修飾状態の情報を考慮して,機械学習やAIによるゲノム編集効率の予測などもさかんに行われるものと考える.
ゲノム編集は,この技術の汎用性からさまざまな応用分野においても大きく期待されている.微生物での機能性物質産生,農水畜産作物の品種改良,創薬や疾患治療(図4)など,ゲノム編集の可能性は無限大である.生物工学は,微生物の有効利用を遺伝子組換え技術を含むさまざまな技術開発を通して進められてきた.長年研究が進められてきた大腸菌,出芽酵母や分裂酵母などのモデル微生物では,HR修復を介して自在な遺伝子改変が可能であり,ゲノム編集の必要性は低いと感じている研究者も多い.しかしゲノム編集などの新規技術は,モデル微生物においても新しい展開を与える可能性があり,応用技術を開発するためには積極的に導入すべきである.一方,多くの産業微生物では,ゲノム情報の解読は次世代シークエンサーによって容易になっているが,遺伝子導入方法が確立されておらず,改変技術を確立するための選抜マーカーなども揃っていない状況も見受けられる.このような場合は,ガイドRNAとCasの複合体であるリボ核タンパク質複合体(ribonucleoprotein:RNP)を直接導入するなどの方法の開発が,一つの有効な方法かもしれない.
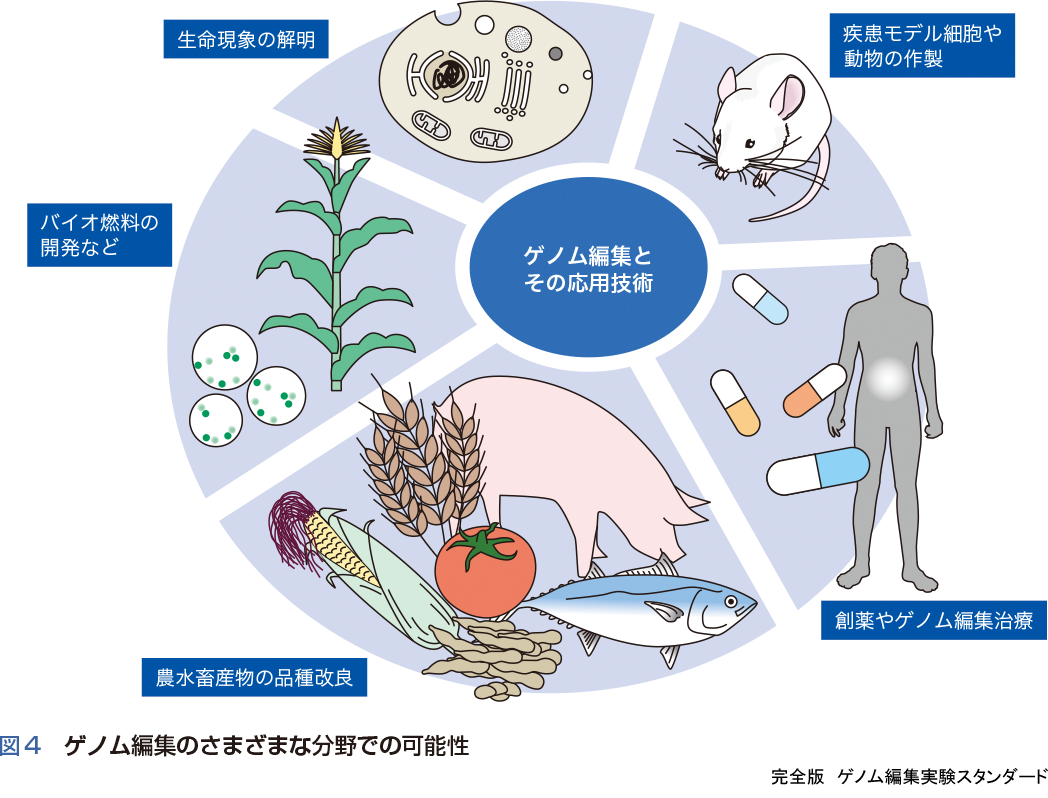
農水畜産分野での品種改良においては,食糧問題を解決するためにゲノム編集は導入を必要とすべき技術の一つであることは間違いない.すでに国内外で遺伝子組換えにあたらないゲノム編集によって複数の作物の作出に成功している.遺伝子組換えにあたらないゲノム編集によって作出した作物の安全性については,食品としての安全性(毒性物質やアレルゲン物質が産生されていないこと)を評価するとともに,環境に与える影響について考察して行く必要がある.
ゲノム編集の医療分野での利用は,応用面で最も期待が大きいと言ってよい.創薬開発用の疾患モデルの細胞や動物作りにゲノム編集技術は不可欠である.例えば,iPS細胞を使った疾患研究や再生医療向け細胞の作製が進められているが,ここでもゲノム編集技術は大きな役割を担う.一塩基レベルでの改変や染色体再編が可能な汎用技術が今後開発されれば,大きなインパクトになると考えられる.ゲノム編集を利用したヒト化哺乳類(香月・押村のコラム)11)や疾患モデル霊長類(Ⅲ-5)12)の作製は,日本が高い技術を有しており,創薬に必要な高いレベルの改変技術が開発されている.ゲノム編集を用いた治療については,生体内ゲノム編集と生体外ゲノム編集によって,利用が進められている.特に,生体外ゲノム編集治療では,がん細胞に対して免疫力を高めた免疫細胞をゲノム編集で作製する研究が海外を中心に進んでいる.中国や米国では,本庶佑博士が発見したPD-1(programmed cell death-1)を破壊した免疫細胞を作製し,これを使ったがん治療の臨床研究をすでに進めている.このような状況から,日本国内においてもゲノム編集治療をめざした研究を進めることが急務であり,早急に研究を進めていく必要がある.
おわりに
ゲノム編集技術は,応用技術を中心に今後さらに大きく発展してくことが予想される.本稿を執筆する間にも,トランスポゼースを利用した技術やprime editingなど注目度の高い開発が次々と報告され,国内においても新しいゲノム編集技術の開発が進んでいる.特に,応用技術の開発は未だ進行中であり,常に最新の情報を収集し,自身の研究に生かしていくことが重要であると感じている.一方,安全面や倫理面でゲノム編集技術の解決すべき問題も浮上しており,研究者のみならず社会学者や市民団体を含めた継続的な議論を続けていくことがますます重要になっている.そのため筆者らは日本ゲノム編集学会(http://jsgedit.jp)において,最新の技術についての情報提供や社会受容活動を進めている.本稿がゲノム編集技術のさらなる開発と利用につながれば幸いである.
文献
- 「ゲノム編集の基礎と応用」(山本 卓/著),裳華房,2018
- Jinek M, et al:Science, 337:816-821, 2012
- Suzuki K, et al:Nature, 540:144-149, 2016
- Strecker J, et al:Science, 365:48-53, 2019
- Tanenbaum ME, et al:Cell, 159:635-646, 2014
- Konermann S, et al:Nature, 517:583-588, 2015
- Shalem O, et al:Science, 343:84-87, 2014
- McKenna A, et al:Science, 353:aaf7907, 2016
- Gootenberg JS, et al:Science, 356:438-442, 2017
- Chen JS, et al:Science, 360:436-439, 2018
- Kazuki Y, et al:Proc Natl Acad Sci U S A, 116:3072-3081, 2019
- Sato K, et al:Cell Stem Cell, 19:127-138, 2016
著者プロフィール
- 山本 卓
- 広島大学大学院統合生命科学研究科/広島大学ゲノム編集イノベーションセンター
- 佐久間哲史
- 広島大学大学院統合生命科学研究科